
撰文 | 我的闺蜜老红帽
#肿瘤#
微生物组在维持人类健康和疾病发生发展过程中具有至关重要的作用,这其中也包括肿瘤。虽然健康的胰腺是否栖息着微生物仍旧不得而知,但是越来越多的证据证实,进入胰腺的细菌和真菌是胰管腺癌(pancreatic ductal adenocarcinoma)的元凶之一【1,2】。菌群及其代谢产物可以改变基因表达水平,从而导致DNA损伤【3】,激活模式识别受体【4】,诱导炎症以及免疫抑制作用【5】。菌群同样参与胰管腺癌病患对药物等治疗方式的耐受作用,包括菌群胞苷脱氢酶(microbial cytidine deaminase)可以使得吉西他滨(gemcitabine)失活【6】,而抗生素导致的肠道菌群衰减可以增强机体对免疫检查点抑制剂的敏感性【7】。胰管腺癌幸存者的肿瘤样本中往往菌群种类更为丰富,并且,肿瘤抗原往往与微生物多肽同源【8】。
尽管胰腺肿瘤菌群的研究已经初具规模,但是仍有若干难题横亘其中。第一,许多肠道菌群难以在体外培养。第二,菌群组分比例的个体差异性很大,并且,基本上没有动物模型可以很好的模拟人类肿瘤-菌群相互作用【9】。第三,术后样本污染使得菌群数据可信性降低。近期研究已经通过对数百种人类原发肿瘤进行16S细菌核糖体DNA测序的方法,构建了肿瘤类型特异的菌群特征标签。当然,这些从组织而来的基因组学数据并没有进行细菌-体细胞富集,也没有细胞类型特异性活化以及微生物-细胞相互作用的信息。尤其值得注意的是,胰管腺癌的病理特征之一是间质纤维化,它们占据了胰腺肿瘤的绝大部分体积,这样从组织测序数据中探寻细胞联系的难度就进一步增加。
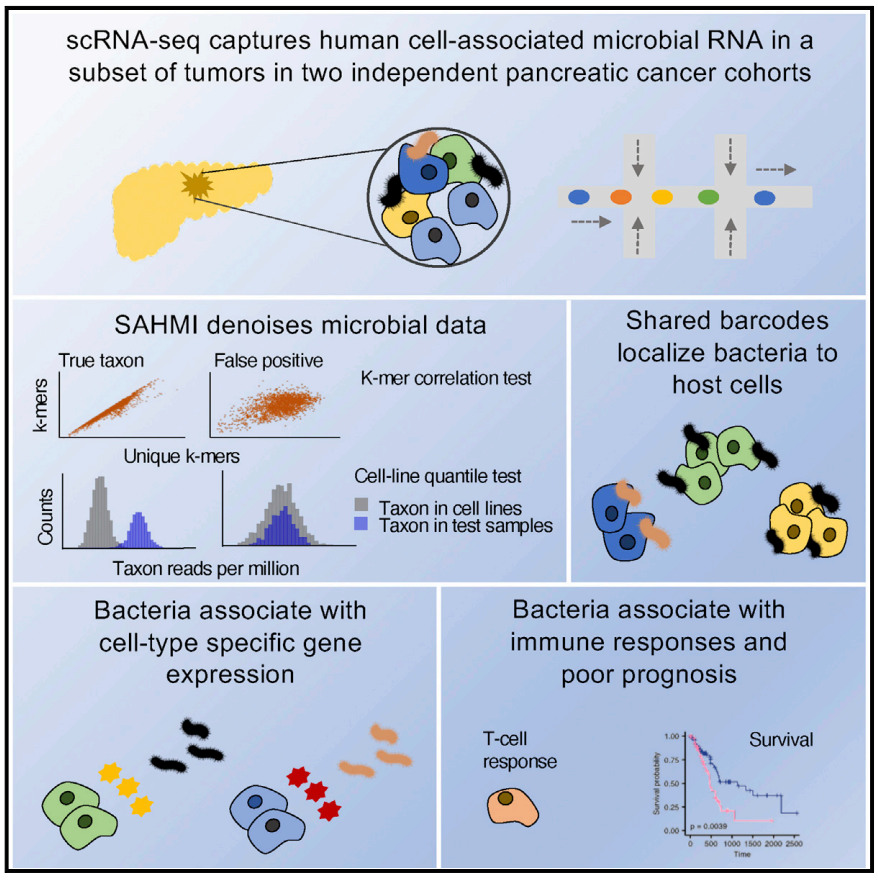
近日,来自美国Rutgers University 的Martin J. Blaser 和Subhajyoti De研究组在Cancer Cell上发表题为Tumor microbiome links cellular programs and immunity in pancreatic cancer的文章,通过对宿主-菌群相互作用进行单细胞分析(single-cell analysis of host-microbiome interactions),研究了胰腺肿瘤的肿瘤-微生物相互作用。

作者近期建立的“宿主-菌群相互作用单细胞分析系统”,可以在单细胞尺度对人类临床组织样本进行宿主-菌群相互作用分析。为了检测这一系统的稳定性和准确性,作者采集分析了4组来源于单细胞RNA测序和全基因组测序的肿瘤数据,以及14组来源于RNA测序和16S核糖体DNA测序的肿瘤和瘤旁组织数据。确定通过上述系统,这些数据均可以检测到细菌信息;还通过独立的两组大规模胰腺管癌单细胞RNA测序数据(scPDA1和scPDA2)进一步确定上述结论。
接下来,作者从scPDA1和scPDA2的55份胰腺样本中检测到48份(87%)含有菌群信息,其中,两组样本共同检测到19个属的细菌。这些菌通常都与肠道疾病相关。含量最为丰富的是Campylobacter spp(弯曲菌),它可以造成肠道或者全身炎症。与其它类型的细胞相比,肿瘤细胞与细菌相互作用最为明显。这也在单细胞水平说明在肿瘤微环境中,细菌与肿瘤细胞活动相关,而且,肿瘤中的细菌可以诱导免疫细胞浸润以及抗肿瘤免疫响应活化。而且,胰腺管癌肿瘤微环境中的T细胞转录谱与感染环境更为类似,这也在一定程度上解释了胰腺肿瘤微环境中的高水平炎症状态的来源,以及免疫疗法疗效不够理想的原因。
最后,作者研究肿瘤微环境中的细菌是否与患者整体生存率相关。因为缺乏临床数据支持,作者通过Cox比例风险回归模型(univariate Cox proportional hazard models)预测,胰腺肿瘤患者的生存率与细菌具有一定相关性。
综上所述,作者建立了检测胰腺肿瘤微生物组的宿主-菌群相互作用单细胞分析系统,并发现肿瘤微环境中的细菌与肿瘤、免疫响应以及患者生存率均相关。
原文链接:
https://doi.org/10.1016/j.ccell.2022.09.009
参考文献
1. Vitiello, G.A., Cohen, D.J., and Miller, G. (2019). Harnessing the microbiome for pancreatic cancer immunotherapy. Trends Cancer5, 670–676.
2. Wei, M.Y., Shi, S., Liang, C., Meng, Q.C., Hua, J., Zhang, Y.Y., Liu, J., Zhang, B., Xu, J., and Yu, X.J. (2019). The microbiota and microbiome in pancreatic cancer: more influential than expected. Mol. Cancer 18, 97.
3. O ̈ g!rendik, M. (2017). Periodontal pathogens in the etiology of pancreatic can- cer. Gastrointest. Tumors3, 125–127.
4. Ochi, A., Nguyen, A.H., Bedrosian, A.S., Mushlin, H.M., Zarbakhsh, S., Barilla, R., Zambirinis, C.P., Fallon, N.C., Rehman, A., Pylayeva-Gupta, Y., et al. (2012). MyD88 inhibition amplifies dendritic cell capacity to promote pancre- atic carcinogenesis via Th2 cells. J. Exp. Med. 209, 1671–1687.
5. Aykut, B., Pushalkar, S., Chen, R., Li, Q., Abengozar, R., Kim, J.I., Shadaloey, S.A., Wu, D., Preiss, P., Verma, N., et al. (2019). The fungal mycobiome pro- motes pancreatic oncogenesis via activation of MBL. Nature574, 264–267.
6. Geller, L.T., Barzily-rokni, M., Danino, T., Jonas, O.H., Shental, N., Nejman, D., Gavert, N., Zwang, Y., Cooper, Z.A., Shee, K., et al. (2017). Potential role of in- tratumor bacteria in mediating tumor resistance to the chemotherapeutic drug gemcitabine. Science357, 1156–1160.
7. Pushalkar, S., Hundeyin, M., Daley, D., Zambirinis, C.P., Kurz, E., Mishra, A., Mohan, N., Aykut, B., Usyk, M., Torres, L.E., et al. (2018). The pancreatic can- cer microbiome promotes oncogenesis by induction of innate and adaptive immune suppression. Cancer Discov.8, 403–416.
8. Balachandran, V.P., quksza, M., Zhao, J.N., Makarov, V., Moral, J.A., Remark, R., Herbst, B., Askan, G., Bhanot, U., Senbabaoglu, Y., et al. (2017). Identification of unique neoantigen qualities in long-term survivors of pancre- atic cancer. Nature551, 512–516.
9. Mallapaty, S. (2017). Gnotobiotics: getting a grip on the microbiome boom. Lab Anim. (NY) 46, 373–377.


想了解更多精彩内容,快来关注BioArt生物艺术
| 留言与评论(共有 0 条评论) “” |